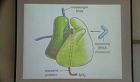
인간면역결핍바이러스 게놈RNA의 바이러스내부로의 encapsidation 과정은 바이러스 생활 주기에서 필수적인 과정이다. 이러한 게놈 RNA 의 encapsidation 은 gag 중합효소내의 NC 도메인과 게놈 RNA ...
http://chineseinput.net/에서 pinyin(병음)방식으로 중국어를 변환할 수 있습니다.
변환된 중국어를 복사하여 사용하시면 됩니다.
- 中文 을 입력하시려면 zhongwen을 입력하시고 space를누르시면됩니다.
- 北京 을 입력하시려면 beijing을 입력하시고 space를 누르시면 됩니다.
인간면역결핍바이러스 제1형(HIV-1)의 nucleocapsid 단백질에 결합하는 RNA 의 시험관내 선택 및 세포내 특성분석 = In vitro selection and charicterization of high affinity RNA to nucleocapsid protein of HIV-1
한글로보기https://www.riss.kr/link?id=T8421474
- 저자
-
발행사항
서울 : 단국대학교 대학원, 2000
-
학위논문사항
학위논문(석사) -- 단국대학교 대학원 , 생물학과 동물학전공 , 2000. 8
-
발행연도
2000
-
작성언어
한국어
- 주제어
-
KDC
491.29 판사항(4)
-
발행국(도시)
서울
-
형태사항
45p. ; 26cm.
- 소장기관
-
0
상세조회 -
0
다운로드
부가정보
국문 초록 (Abstract)
인간면역결핍바이러스 게놈RNA의 바이러스내부로의 encapsidation 과정은 바이러스 생활 주기에서 필수적인 과정이다.
이러한 게놈 RNA 의 encapsidation 은 gag 중합효소내의 NC 도메인과 게놈 RNA 상의 RNA packaging 신호인 Psi 염기서열간의 특이적인 상호작용에의해 이루어진다.
NC단백질은 basic한 아미노산과 두 개의 zinc finger motif를 포함한다. 이러한 특징은 게놈 RNA의 packaging 에 영향을 줄것으로 보인다.
Psi 염기서열은 게놈 RNA 의 5' 말단에 위치하는 부위이며 4개의 stem-loop 구조를 형성한다.
Nucelocapsid 단백질의 Psi 염기서열 결합은 RNA packaging과 감염성 바이러스 생성에 중요한 역할을 담당한다.
우리는 이러한 상호작용을 저해시키기위해 nucelocapsid 단백질에대한 친화력 높은 RNA를 선별하였다. SELEX(Systematic Evolution of Ligans by EXponential enrichment)는 시험관내 선별 방법으로 무작위적인 RNA pool로부터 목표분자에 가장 특이적으로 결합하는 물질을 분리해내는 방법이다.
이 논문에서는 NC단백질에 대한 친화력 높은 RNA를 분리해내었다.
NC단백질에 결합하는 RNA 는 10^13의 RNA 복합성 라이브러리로부터 선택과 증폭 단계를 반복함으로서 분리해내었다. 두 번의 독립적인 선택과정으로 NC단백질에 결합하는 RNA를 분리하는 실험을 수행하였다. 이러한 두 번의 선택과정으로부터 얻은 염기서열은 매우 유사하였다. 이러한 결과는 선택된 RNA가 NC단백질에 매우 특이적임을 나타낸다.
선택한 RNA는 Surface Plasmon Resonance와 gel shift mobility assay를 이용하여 결합정도를 알아보았다.선택한 RNA는 약 Kd =10nM 이하의 결합도를 나타냈으며 original RNA pool보다 50배 높은 친화력이다.
선택한 RNA의 특이성을 알아보기위해 gel shift competition assay를 수행하였다.
그러므로, 선택한 RNA는 NC-Psi의 상호작용을 억제할수 있고 항 HIV 약제로서 사용도 가능할 것이다.
다국어 초록 (Multilingual Abstract)
Encapsidation of HIV-1 genomic RNA into the virion is an essential step of the viral life cycle. Genomic RNA encapsidation is mediated by specific interaction between the nucleocapsid (NC) domain in the gag polyprotein precursor and RNA packaging sig...
Encapsidation of HIV-1 genomic RNA into the virion is an essential step of the viral life cycle. Genomic RNA encapsidation is mediated by specific interaction between the nucleocapsid (NC) domain in the gag polyprotein precursor and RNA packaging signal (φ) in genomic RNA.
NC proteins include a high content of basic residues and two of zinc finger motifs. These features of the NC proteins seem to affect a packaging of genomic RNA.
Psi (φ) sequence is a region at the 5'-end of the genomic RNA and includes four stem-loops structure.
Nucleocapsid protein binding to Psi (φ) sequence plays a critical role in packaging and formation of infectious virions.
To provent these interaction, we used SELEX to selected a high affinity RNA for nucleocapsid protein. SELEX(Systematic Evolution of Ligans by EXponential enrichment ) is an in vitro selection method that has been used to isolate most specific binder for target molecules from randonized RNA pool.
Here se isolated the high afinity RNA for the nucleocapsid protein.
RNAs that binds to NC protein were isolated from combinatorial library of 10^13 RNA molecules by selction - amplification cycles.
The two independent selection experiments were done to selec RNAs that bind to the NC proteins.
Sequences from these two selection experiments are very similar.
The result implies that selected RNAs bind very specific for NC proteins.
selected RNAs were tested for binding to NC protein by Surface Plasmon Resonance and gel shift mobility assay.
목차 (Table of Contents)
- 1. 서론 = 1
- 2. 실험재료 및 방법
- 2.1 SELEX
- 2.1.1 DNA 라이브러리의 고안과 합성 = 4
- 2.1.2 RNA 라이브러리의 합성 = 4
- 1. 서론 = 1
- 2. 실험재료 및 방법
- 2.1 SELEX
- 2.1.1 DNA 라이브러리의 고안과 합성 = 4
- 2.1.2 RNA 라이브러리의 합성 = 4
- 2.1.3 시험관내 선택 = 5
- (1) selection과정
- (2) 역전사PCR 및 전사
- 2.2 Precipitation assay
- 2.2.1 [α-32p]UTP를 사용한 RNA 표지 = 7
- 2.2.2 Binding assay = 8
- 2.3 Cloning 및 염기서열 분석
- 2.3.1 Cloning = 8
- (1) 제한효소 반응 및 ligation
- (2) 형질전환
- 2.3.2 염기서열분석 = 10
- 2.4 Surface Plasmon Resonance(SPR) 분석 = 10
- 2.5 Gel shift assay
- 2.5.1 NC 단백질에 대한 Kd 측정 = 11
- 2.5.2 Competition assay = 12
- 3. 결과
- 3.1 DNA 라이브러리의 고안과 합성 = 13
- 3.2 RNA 라이브러리의 합성 = 13
- 3.3 시험관내 선택 (SELEX) = 13
- 3.4 Precipitation assay = 14
- 3.5 염기서열 분석 및 RNA 구조 예상 = 14
- 3.6 Surface Plasmon Resonance(SPR) 분석 = 15
- 3.7 Gel shift competition assay에 의한 특이적 결합 측정 = 16
- 3.8 Gel shift에 의한 결합 친화도 분석 = 16
- 4. 결론 및 토의 = 18
- 5. 참고문헌 = 37
- 6. List of Figures = 41
- 7. List of Tables = 42
- 8. Abstract = 43